Unidad 5.1
dgonzalez

Regresión Lineal Simple
Inicialmente los datos son representados en un diagrama de dispersión que ayuda a visualizar el tipo de relación que hay entre ellos
x=c(24.3, 12.5, 31.2, 28.0, 35.1, 10.5, 23.2, 10.0, 8.5, 15.9, 14.7, 15.0)
y=c(16.2, 8.5, 15.0, 17.0, 24.2, 11.2, 15.0, 7.1, 3.5, 11.5, 10.7, 9.2)
plot(x,y, pch=19, col=c5, las=1)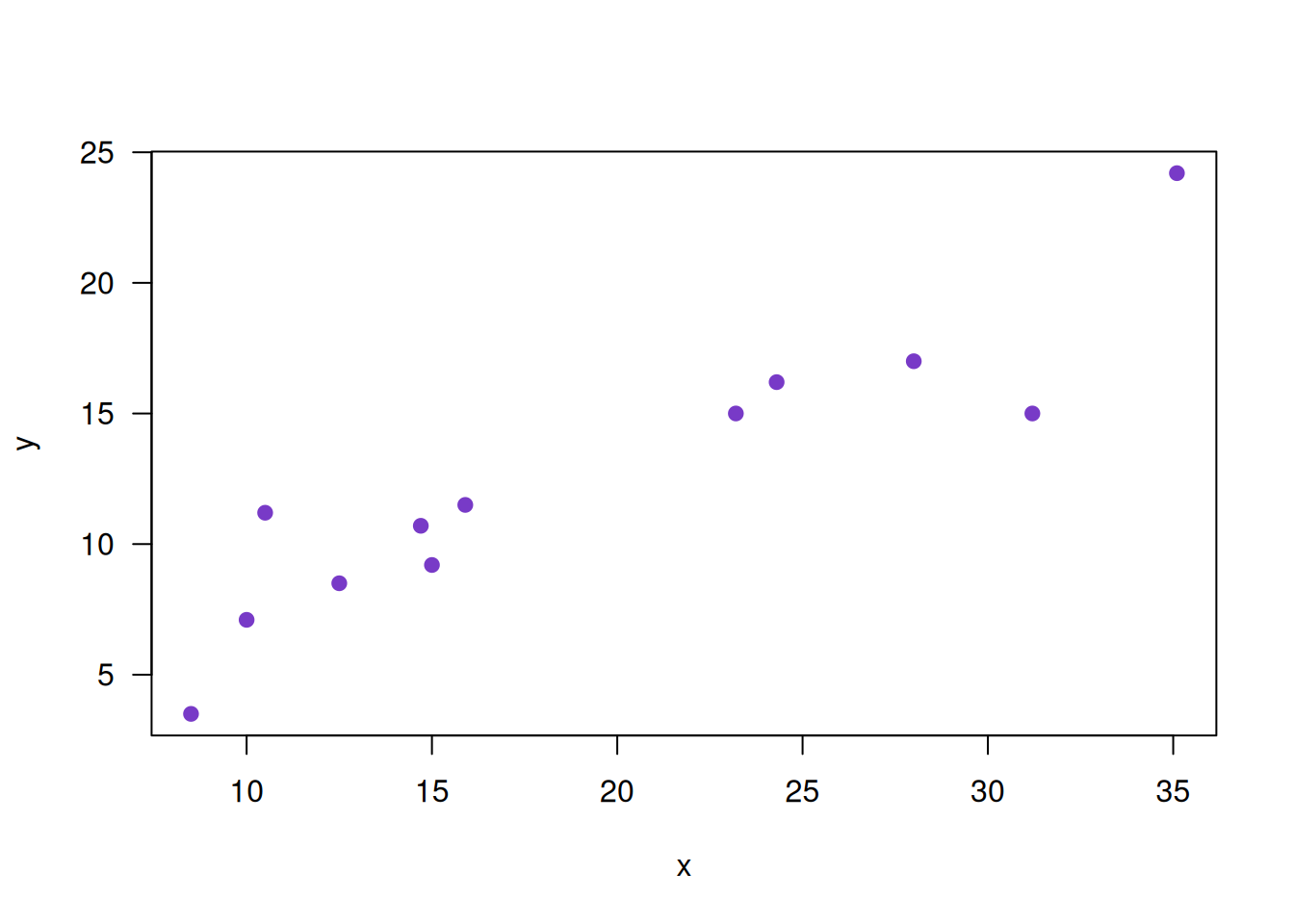
Estimación MCO
Se pretende estimar un modelo que represente los datos mediante una linea recta
\(\widehat{y}= b_{0} + b_{1} x\)
Donde + \(b_{0}\) : representa el intercepto + \(b_{1}\) : representa la pendiente
la función utilizada para la estimación de los parámetros por el método de MCO es : lm()
regresion=lm(y ~ x)
plot(x,y, xlab = "Ingresos", ylab = "Consumo estimado", pch=19, col=c3, las=1)
abline(regresion)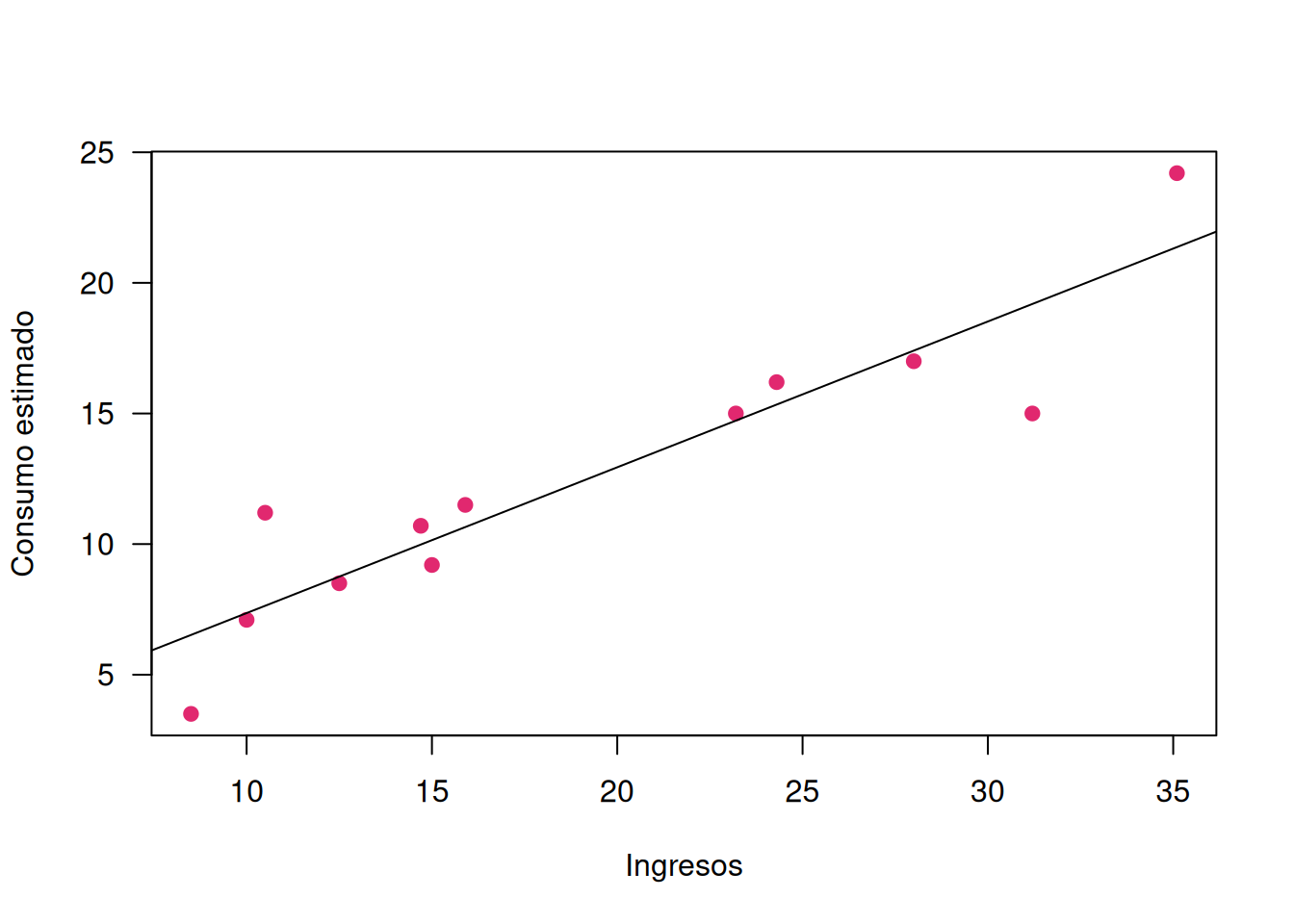
uhat=regresion$residuals
yhat=regresion$fitted.values
data.frame(y,x,yhat, uhat) y x yhat uhat
1 16.2 24.3 15.341446 0.8585544
2 8.5 12.5 8.755023 -0.2550230
3 15.0 31.2 19.192828 -4.1928284
4 17.0 28.0 17.406680 -0.4066799
5 24.2 35.1 21.369697 2.8303031
6 11.2 10.5 7.638680 3.5613199
7 15.0 23.2 14.727457 0.2725429
8 7.1 10.0 7.359594 -0.2595944
9 3.5 8.5 6.522337 -3.0223373
10 11.5 15.9 10.652806 0.8471942
11 10.7 14.7 9.983000 0.7169999
12 9.2 15.0 10.150451 -0.9504515Validación de supuestos
La verificación de los supuestos constituye un requisito excencial para la realización de inferencia sobre los parámetros estimados
Supuesto 1
Normalidad de los errores
library(fBasics)
# Test de normalidad de los errores
# Ho: los errores tienen distribución normal
ks.test(uhat, "pnorm") # test de normalidad
Exact one-sample Kolmogorov-Smirnov test
data: uhat
D = 0.17998, p-value = 0.7698
alternative hypothesis: two-sidedshapiro.test(uhat) # test de normalidad
Shapiro-Wilk normality test
data: uhat
W = 0.94569, p-value = 0.5751# se debe instalar paquete fBasics
# install.packages("fBasics")
jarqueberaTest(uhat) # test de normalidad
Title:
Jarque-Bera Normality Test
Test Results:
STATISTIC:
X-squared: 0.2039
P VALUE:
Asymptotic p Value: 0.9031 Supuesto 2
Modelo completo, esto se traduce en que el término error tiene media cero.
# t-test para verificar E[u]=0, modelo completo
# Ho: miu_u = 0
t.test(uhat)
One Sample t-test
data: uhat
t = -5.9676e-17, df = 11, p-value = 1
alternative hypothesis: true mean is not equal to 0
95 percent confidence interval:
-1.363421 1.363421
sample estimates:
mean of x
-3.696678e-17 Supuesto 3
Los errores no estan autocorrelacionados, es decir son independientes unos de otros
# Test de Autocorrelacion de errores
# Cor[ui,uj]=0
# Ho: no existe autocorrelacion entre los errores
# se debe instalar paquete lmtest
# install.packages("lmtest")
library(lmtest) #dwtest
dwtest(y ~ x) # test de Durbin-Wapson
Durbin-Watson test
data: y ~ x
DW = 1.5517, p-value = 0.1769
alternative hypothesis: true autocorrelation is greater than 0Supuesto 4
Los errores tienen varianza constante
# Test de Homoscedasticidad
# Ho: la varianza de los erroes es constante
# Ho: V(u) = sigma2
gqtest(y ~ x)
Goldfeld-Quandt test
data: y ~ x
GQ = 0.19285, df1 = 4, df2 = 4, p-value = 0.93
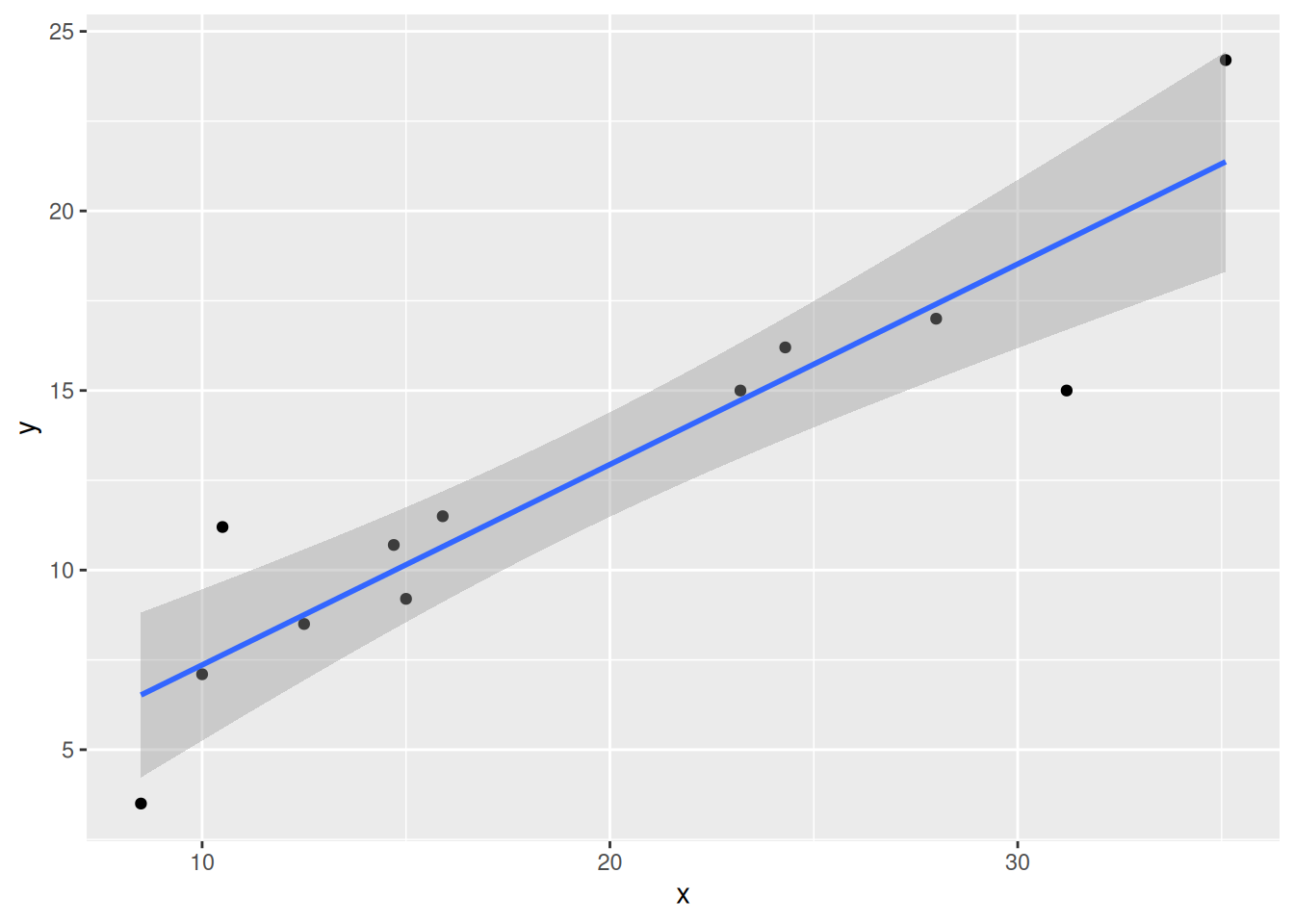
alternative hypothesis: variance increases from segment 1 to 2library(ggplot2)
x=c(24.3, 12.5, 31.2, 28.0, 35.1, 10.5, 23.2, 10.0, 8.5, 15.9, 14.7, 15.0)
y=c(16.2, 8.5, 15.0, 17.0, 24.2, 11.2, 15.0, 7.1, 3.5, 11.5, 10.7, 9.2)
datos=data.frame(x,y)
p <- ggplot(datos, aes(x, y)) +
geom_point()
p + geom_smooth(method = "lm", level = 0.95)